티스토리 뷰
R | COVID-19 데이터로 무엇을 할 수 있을까 | 3. 전체적인 상황을 볼 수 있는 stacked area plot
Chloe A_Choe 2020. 4. 22. 14:20

지난 포스팅에 준비해둔 데이터를 이용해서 우리나라의 코로나 진행 상황을 한눈에 볼 수 있는 그래프를 그려보려고합니다. 날짜의 흐름에 따라 Active, Cured(Recovered), Death 수를 쌓아서, 점차 나아지는 상황을 시각화 해보겠습니다.
분석에 사용할 데이터
> mydata_tb_Korea <- mydata_tb %>%
subset(mydata_tb$Country=='Korea, South')
> mydata_tb_Korea %>% tail
Date Country Confirmed Recovered Deaths Date.no Active Confirmed.new.day Recovered.new.day Deaths.new.day Confirmed.new.week Recovered.new.week Deaths.new.week
8185 2020-04-15 Korea, South 10591 7616 225 85 2750 27 82 3 168 643 21
8186 2020-04-16 Korea, South 10613 7757 229 86 2627 22 141 4 163 640 21
8187 2020-04-17 Korea, South 10635 7829 230 87 2576 22 72 1 155 586 19
8188 2020-04-18 Korea, South 10653 7937 232 88 2484 18 108 2 141 569 18
8189 2020-04-19 Korea, South 10661 8042 234 89 2385 8 105 2 124 595 17
8190 2020-04-20 Korea, South 10674 8114 236 90 2324 13 72 2 110 580 14먼저 데이터에서 한국 데이터를 subset하도록 합니다.
subset
> mydata_tb_Korea_long <- melt(mydata_tb_Korea[,c('Date', 'Active', 'Recovered', "Deaths")])
> mydata_tb_Korea_long %>% head
Date variable value
1 2020-01-22 Active 1
2 2020-01-23 Active 1
3 2020-01-24 Active 2
4 2020-01-25 Active 2
5 2020-01-26 Active 3
6 2020-01-27 Active 4
> mydata_tb_Korea_long %>% str
'data.frame': 270 obs. of 3 variables:
$ Date : chr "2020-01-22" "2020-01-23" "2020-01-24" "2020-01-25" ...
$ variable: Factor w/ 3 levels "Active","Recovered",..: 1 1 1 1 1 1 1 1 1 1 ...
$ value : int 1 1 2 2 3 4 4 4 4 11 ...현재 데이터는 wide 형태인데 melt()를 이용해서 long format 으로 변경을 해주어야합니다. 이때 필요한 데이터 Date, Active, Recovered, Deaths 만 추려줍니다. melt에 관련한 포스팅은 여기
geom_bar
> ggplot(mydata_tb_Korea_long, aes(x=Date, y=value, fill=variable)) +
+ geom_bar(position="stack", stat="identity") +
+ scale_fill_viridis(discrete = T) +
+ theme_minimal()
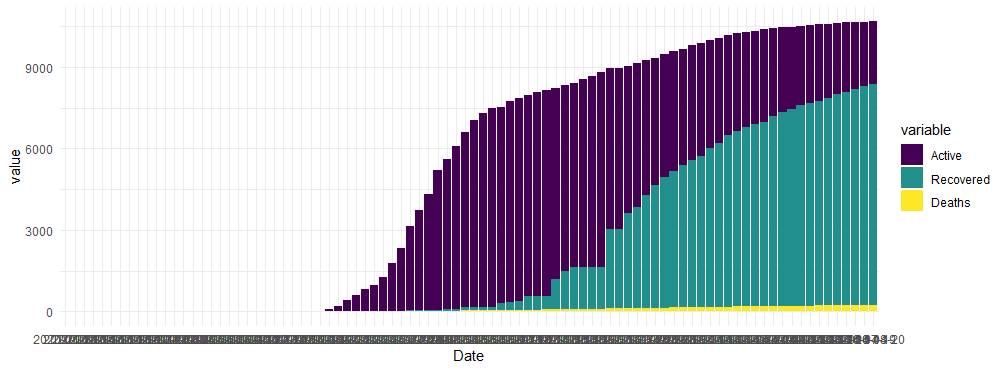
ggplot 에서 geom_bar를 이용해서 한번 그래프를 그려보았습니다. 수정을 해야할 곳이 몇군데 보이는데요.
1. 쌓아올려진그래프를 위해 position='stack'으로 하였지만 결과로 나온 그래프를 보니 Active Recovered, Deaths의 순서가 반대로 바꿔줘야 합니다. 순서가 반대로 있으니 너무 이상한 그래프네요.
2. 그리고, x축의 날짜 정보를 보니 Date 값으로 인식을 못하고 그저 글자로 인식하고있네요. 이것도 수정을 해줘야 하겠습니다.
3. 마지막으로는 geom_bar대신에 geom_area로 바꿔주면 좀더 매끄러운 그래프를 얻을 수 있을 것 같습니다.
수정 1. variable level 변경
> mydata_tb_Korea_long $variable <- factor(mydata_tb_Korea_long$variable, levels=c("Deaths", "Recovered", "Active"))
> mydata_tb_Korea_long $variable %>% levels()
[1] "Deaths" "Recovered" "Active" factor를 이용해서 level을 새로 지정해줍니다. variable의 levels가 변경된것을 확인 할 수 있네요.
수정 2. Date 형식 변경
> install.packages('lubridate')
> library(lubridate)
우선 lubridate 패키지가 없다면 설치를 하고, 라이브러리를 불러옵니다.
> mydata_tb_Korea_long$Date <- ymd(mydata_tb_Korea_long$Date)
> mydata_tb_Korea_long %>% str
'data.frame': 270 obs. of 3 variables:
$ Date : Date, format: "2020-01-22" "2020-01-23" ...
$ variable: Factor w/ 3 levels "Deaths","Recovered",..: 3 3 3 3 3 3 3 3 3 3 ...
$ value : int 1 1 2 2 3 4 4 4 4 11 ...그다음 ymd()를 이용해서 date format으로 변경을 해줍니다. 제대로 변경이 된것을 확인 할 수 있습니다.
수정 3. geom_area()
> ggplot(mydata_tb_Korea_long, aes(x=Date, y=value, group=variable, fill=variable)) +
+ geom_area(stat="identity", position='stack') +
+ scale_fill_viridis(discrete = T) +
+ theme_light()
geom_area 그래프로 변경되니 훨씬 깔끔한 모습을 볼 수 있고, x 축 또한 간결하게 정리된 모습을 볼 수 있습니다.
Result
> ggplot(mydata_tb_Korea_long, aes(x=Date, y=value, group=variable, fill=variable)) +
+ geom_area(stat="identity", position='stack') +
+ scale_fill_viridis(discrete = T) +
+ labs(title = sprintf('COVID-19 Cases in South Korea'),
+ subtitle = sprintf('Last update : %s', Date_update),
+ fill = 'Cases',
+ caption = 'made by Chloe',
+ y = 'Number of Cases',
+ x = 'Date') +
+ theme_light()

한국의 코로나바이스 진행상황을 한번에 파악할 수 있는 그래프로 시각화해보았습니다. 현재의 상태로 계속 진행되길 바라며, 여름 전에는 안정화된 한국의 모습을 기대해봅니다.
다른 나라들은 현재 어떤 모습일까?
CH
중국의 경우는 안정화에 들어선 모습과 함께, 빠른속도로 치료된 환자수도 빠르게 증가한 모습을 볼 수 있습니다.

NL
네덜란드의 경우는 치료된 환자의 수가 거의 없고, 확진환자만 늘어나는 추세가 이상해보였는데, 기사를 찾아보니 치료된 환자의 수를 제공하고 있지 않다고 합니다. 치료된 환자의 추세를 제공하지 않는 국가의 경우에는 현재 시각화가 전혀 도움이 되지 않네요.

확진자수가 가장 많은 다섯 국가의 데이터
확진자수가 가장 많은 다섯국가중에는 독일만 환자수가 점진적으로 감소하고있는 모습을 보이고 있고, 가장 큰 문제는 미국으로 보입니다.

'R' 카테고리의 다른 글
| R | ceiling()를 이용한 올림, floor()를 이용한 내림 (0) | 2020.05.06 |
|---|---|
| R | round()를 이용한 반올림 (0) | 2020.05.06 |
| R | COVID-19 데이터로 무엇을 할 수 있을까 | 2. 데이터 다운로드와 전처리 (0) | 2020.04.21 |
| R | str_match()를 이용해 원하는 단어 추출하기(stringr) (0) | 2020.04.13 |
| R | COVID-19 데이터로 무엇을 할 수 있을까 | 1. 코로나바이러스감염증이란? (0) | 2020.04.01 |
- Total
- Today
- Yesterday
- hist
- covid
- Heatmap
- r
- Excel
- geom_line
- Visualization
- geom_bar
- visualizing
- 숫자
- coronavirus
- 코로나바이러스
- 엑셀
- Python
- 데이터
- Order
- plot
- comma
- SEQ
- Command
- 파이썬
- Cast
- RStudio
- data
- 팟빵
- Coding
- BIOINFORMATICS
- for loop
- format
- 2진수
| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | |||
| 5 | 6 | 7 | 8 | 9 | 10 | 11 |
| 12 | 13 | 14 | 15 | 16 | 17 | 18 |
| 19 | 20 | 21 | 22 | 23 | 24 | 25 |
| 26 | 27 | 28 | 29 | 30 | 31 |
